Illumina作为当今二代测序市场占比份额最大的测序平台,理解其测序接头的具体结构,有助于我们了解各种建库方法。下面,我就来介绍一下illumina的接头结构以及基于此衍生出的设计模式。
Y型接头
最常规的illumina接头就是俗称Y字形的接头,其结构如下:
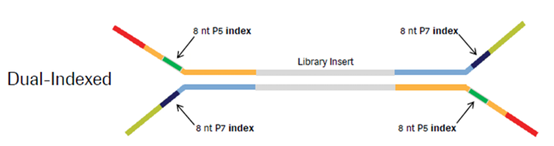
如上图所示,红色和浅绿色的是固定序列,作用是成簇反应(桥式PCR的引物),使得文库DNA分子经过桥式PCR可以“长”到测序芯片上面,因此它的序列是固定的,也称为P5、P7序列,建好的文库使用这对引物扩增,可以起到放大文库量的作用。上述浅黄色和浅蓝色的片段,是测序引物,其作用是分别读取插入片段的两侧,可以理解为序列读取位置的指示。需要说明的是,测序引物还可以作为测定标签分子序列的测序引物使用。深蓝色和深绿色的,分别标注有8nt P7 index和8nt P5 index的区段,就是我们常说的标签分子序列,用于识别不同文库的序列的。
实际上,我们合成的引物都是单链的,illumina推出的接头是双链的。两条单链引物直接仅有几十碱基是互补配对的,其余碱基不会形成互补配对关系。因此,退火好的接头,需要低温保存,且尽量避免反复冻融。
PCR方法建库的接头结构
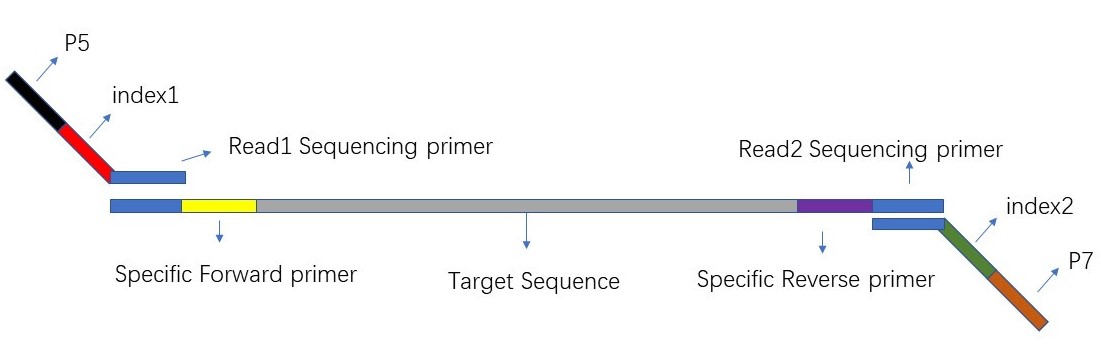
如上图所示的,是扩增获得illumina文库的设计结构。第一轮PCR反应,特异性上下游引物分别携带部分Read1测序引物、Read2测序引物,PCR反应完成之后,即可获得带有部分接头的扩增产物,以此为模板,设计二轮扩增引物,引物与第一轮的Read1测序引物、Read2测序引物部分互补,进行二次扩增,以补齐完整的P5、P7引物,从而获得完整的P5、P7结构。那么就可以在illumina测序仪上进行测序了。需要说明的是,这样的结果也可以简化为一步法,那就是直接合成携带完整P5、P7引物的“特异性”引物,那么PCR反应完成后,经过必要的纯化,即可获得测序文库。该方法的问题在于会提高合成成本,且需要稍微摸索一下扩增反应体系。
小Y字形接头

如上图所示,将illumina原来的大Y字形改短之后,变成小Y字形,通过增加P5、P7端引物长度,从而获得完整illumina文库。别看就这小小的改动,却对大规模建库及避免重复起到了极其重要的作用。本质上说,这种设计结构是将index序列下放到二次扩增的引物上了。这样做的好处是:a)进行DNA连接的时候,不需要对应很多事先准备好的长接头,而是全部采用同一管来源的短接头,操作速度会加快,且不担心交叉污染;b)如果有2组人同时建库的时候,一旦发现index重叠现象,只要还保留有原始的DNA连接产物,即可快速重新组合index,重新扩增,获得新的文库。由于二次扩增循环数普遍是8-12个左右,因此时间上来讲,半个小时到45分钟即可解决问题,不会耽搁某一组的实验进程。c)index引物不需要退火,合成单链就行,降低合成成本。
5. UMI接头
随着illumina推出NovaSeq6000机器以来,极大提高了测序通量。这同时带来了一个新的问题,就是测序的过程中,容易发生index读取错误,尤其是index引物没有纯化干净的时候更为明显。另外,随着对极低频突变检测的需求,需要确保PCR扩增中引入的错误被排除,因此IDT公司开发出了UDI和UMI甚至合二为一的接头。其结构如上图所示。与之前的接头的区别主要是2点:第一,P5、P7两端的index均是唯一,不再交叉组合,可以最大限度避免污染,从而导致数据搞错的现象;第二,P7端index后面还接了一段UMI分子,其实就是一段随机合成的“N”碱基,长度最高是16个碱基。不过,由于测序供应商不倾向于测这类模式,因此这段UMI可以设计在P7端的插入片段位置,那样对于合成短接头的要求就极高,这就是另外一个课题了。
Illumina作为当今二代测序市场占比份额最大的测序平台,理解其测序接头的具体结构,有助于我们了解各种建库方法。下面,我就来介绍一下illumina的接头结构以及基于此衍生出的设计模式。
Y型接头
最常规的illumina接头就是俗称Y字形的接头,其结构如下:

如上图所示,红色和浅绿色的是固定序列,作用是成簇反应(桥式PCR的引物),使得文库DNA分子经过桥式PCR可以“长”到测序芯片上面,因此它的序列是固定的,也称为P5、P7序列,建好的文库使用这对引物扩增,可以起到放大文库量的作用。上述浅黄色和浅蓝色的片段,是测序引物,其作用是分别读取插入片段的两侧,可以理解为序列读取位置的指示。需要说明的是,测序引物还可以作为测定标签分子序列的测序引物使用。深蓝色和深绿色的,分别标注有8nt P7 index和8nt P5 index的区段,就是我们常说的标签分子序列,用于识别不同文库的序列的。
实际上,我们合成的引物都是单链的,illumina推出的接头是双链的。两条单链引物直接仅有几十碱基是互补配对的,其余碱基不会形成互补配对关系。因此,退火好的接头,需要低温保存,且尽量避免反复冻融。
PCR方法建库的接头结构

如上图所示的,是扩增获得illumina文库的设计结构。第一轮PCR反应,特异性上下游引物分别携带部分Read1测序引物、Read2测序引物,PCR反应完成之后,即可获得带有部分接头的扩增产物,以此为模板,设计二轮扩增引物,引物与第一轮的Read1测序引物、Read2测序引物部分互补,进行二次扩增,以补齐完整的P5、P7引物,从而获得完整的P5、P7结构。那么就可以在illumina测序仪上进行测序了。需要说明的是,这样的结果也可以简化为一步法,那就是直接合成携带完整P5、P7引物的“特异性”引物,那么PCR反应完成后,经过必要的纯化,即可获得测序文库。该方法的问题在于会提高合成成本,且需要稍微摸索一下扩增反应体系。
小Y字形接头

如上图所示,将illumina原来的大Y字形改短之后,变成小Y字形,通过增加P5、P7端引物长度,从而获得完整illumina文库。别看就这小小的改动,却对大规模建库及避免重复起到了极其重要的作用。本质上说,这种设计结构是将index序列下放到二次扩增的引物上了。这样做的好处是:a)进行DNA连接的时候,不需要对应很多事先准备好的长接头,而是全部采用同一管来源的短接头,操作速度会加快,且不担心交叉污染;b)如果有2组人同时建库的时候,一旦发现index重叠现象,只要还保留有原始的DNA连接产物,即可快速重新组合index,重新扩增,获得新的文库。由于二次扩增循环数普遍是8-12个左右,因此时间上来讲,半个小时到45分钟即可解决问题,不会耽搁某一组的实验进程。c)index引物不需要退火,合成单链就行,降低合成成本。
5. UMI接头
随着illumina推出NovaSeq6000机器以来,极大提高了测序通量。这同时带来了一个新的问题,就是测序的过程中,容易发生index读取错误,尤其是index引物没有纯化干净的时候更为明显。另外,随着对极低频突变检测的需求,需要确保PCR扩增中引入的错误被排除,因此IDT公司开发出了UDI和UMI甚至合二为一的接头。其结构如上图所示。与之前的接头的区别主要是2点:第一,P5、P7两端的index均是唯一,不再交叉组合,可以最大限度避免污染,从而导致数据搞错的现象;第二,P7端index后面还接了一段UMI分子,其实就是一段随机合成的“N”碱基,长度最高是16个碱基。不过,由于测序供应商不倾向于测这类模式,因此这段UMI可以设计在P7端的插入片段位置,那样对于合成短接头的要求就极高,这就是另外一个课题了。

 举报
举报





 举报
举报

